DNA-Chip-basierte Erreger- und Resistenzdiagnostik
18.01.2014 -
DNA-Chip-basierte Erreger- und Resistenzdiagnostik. Multiresistente Bakterien stellen sowohl für Krankenhäuser als auch außerhalb eine große Bedrohung dar.
Sie sind die wichtigste Ursache für das Behandlungsversagen von Infektionskrankheiten und von daher von großer Bedeutung für Ärzte und Pharmaindustrie. Während der letzten Jahrzehnte musste weltweit ein kontinuierlicher Anstieg im Auftreten multiresistenter Erreger festgestellt werden.
In der klinischen Praxis hat der intensive Einsatz von Antibiotika auf Intensivstationen oder zur Behandlung immunsuppremierter Patienten zu einer Selektion multiresistenter Erreger geführt.
Das zunehmende Auftreten multiresistenter Bakterien und der Nachweis von Resistenzübertragungen von einem Organismus zum anderen haben zu einem Anstieg nosokomialer Erreger geführt, zu deren Behandlung kaum noch wirksame Antibiotika zur Verfügung stehen.
P. aeruginosa, der häufigste Erreger nosokomialer Pneumonien, besitzt ein ausgeprägtes Potential zur Entwicklung einer Multiresistenz, so dass zunehmend auf Polimyxine wie z.B. Colistin als letzte verbleibende Therapieoption zurückgegriffen werden muss, ungeachtet ihrer Toxizität und Nebenwirkungen.
Betrachtet man die weltweit verzeichnete Zunahme multiresistenter P. aeruginosa Isolate, so benötigt man dringend neue Strategien im Umgang mit dieser Bedrohung.
Standardmethoden in mikrobiologischen Laboren benötigen zur Erreger- und Antibiotikaresistenzidentifizierung zwischen 48 und 72 Stunden, abhängig vom Wachstumsverhalten des jeweiligen Erregers. Ferner deutet der relativ hohe Prozentsatz des Therapieversagens bei P. aeruginosa- Infektionen auf eine höhere Prävalenz von genetisch basierten Resistenzdeterminanten hin, als phänotypisch bestimmt werden kann.
Ein Ansatz zur Lösung des Problems besteht in der Entwicklung molekularbiologischer, schnellerer Methoden zur Erreger- und Antibiotikaresistenzidentifizierung, welche eine frühere und adäquatere Behandlung besonders für Patienten in einem kritischen Zustand erlauben.
Das Robert-Bosch-Krankenhaus hat dafür einen DNA-Chip zur Antibiotikaresistenz- und Virulenzfaktorbestimmung in Pseudomonas aeruginosa entwickelt.
DNA-Chips stellen eine neue technologische Plattform dar und ermöglichen die parallele Identifizierung von Antibiotikaresistenz und Virulenz vermittelnden Determinanten.
Zusätzlich liefert sie als eine auf dem Genotyp basierende Methode epidemiologische Informationen, welche zur Überwachung der Verbreitung von Resistenzmechanismen und Anpassung von Hygienemassnahmen herangezogen werden können.
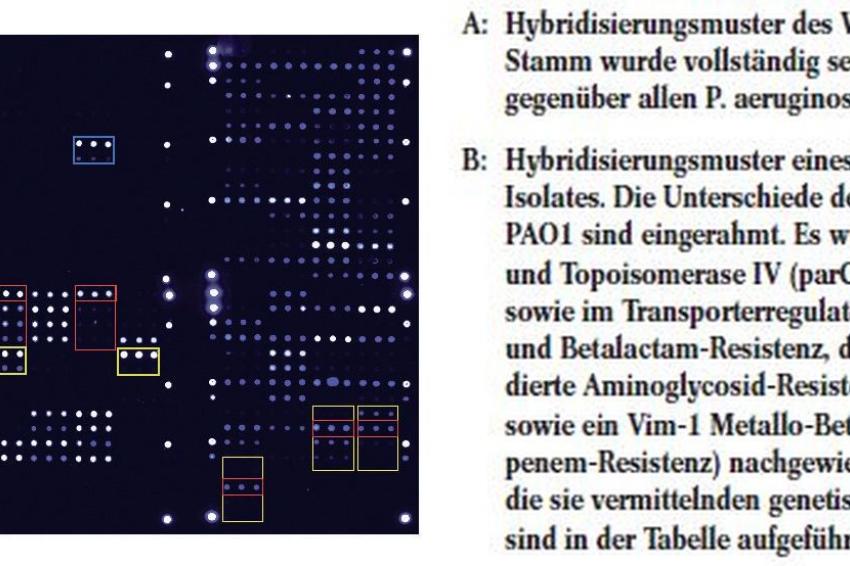
Der entwickelte DNA-Chip deckt Resistenz- vermittelnde Mutationen ungeachtet ihrer Natur (Punktmutationen, Insertionen, Deletionen) in Regulatorgenen (mexR, nfxB, mexT) von Transportsystemen (mexABOprM, MexCD-OprJ, MexEF-OprN), Gyrase (gyrA) und Topoisomerase IV (parC) ab.
Des Weiteren können plasmidkodierte Resistenzgene wie Beta- Laktamasen (imp, vim, oxa, pse, carb) und Aminoglykosid-modifizierende Enzyme (aac, aad, aph) detektiert werden, ebenso Virulenzfaktoren wie TypIII Sekretionstoxine (exoU, exoS, exoT) und Mutationen im mucA Gen.
MucA ist ein Regulator der Alginat Biofilmsynthese in P. aeruginosa und für den mucoiden Phänotyp verantwortlich, eine häufig bei Cystischer Fibrose und Fremdkörper- assoziierten P. aeruginosa-Infektionen beobachtete Phänotypvariante, welche einer antibiotischen Behandlung nur schwer zugänglich ist.
Zusätzlich weist der DNA-Chip Sonden (oprI, gyrB, srv3) für eine eindeutige Speziesidentifizierung von P. aeruginosa auf.
Der entwickelte DNA-Chip besteht aus 202 aminomodifizierten Oligonukleotidsonden (17–24 Nukleotide), welche kovalent an Epoxyd-beschichteten Glasträgern immobilisiert wurden.
Die der PCR und Chip-Hybridisierung (Beispiel in Abb. 1) vorgeschaltete DNAIsolierung erfolgt direkt aus klinischem Material (Blut, Abstriche, BAL, Trachealsekret, etc.).
Vier Multiplex- PCRs wurden zur Amplifikation und gleichzeitigen Fluoreszenzmarkierung durch Einbau von Cy3- dCTP von 66 verschiedenen Sequenzdeterminanten etabliert.
Die beachtliche Verminderung des Zeitbedarfs für die gesamte Prozedur von fünf Stunden verglichen mit 48 bis 72 Stunden bei konventionellen Methoden stellt eine bedeutende Verbesserung dar und ermöglicht gegebenenfalls eine gezieltere Therapie besonders für Patienten auf Intensivstationen.
Eine verbesserte Diagnostik scheint im Augenblick der einzig gangbare Weg zu sein, um das Problem der vermehrt auftretenden multiresistenten Erreger in Angriff zu nehmen, da in den letzten Jahren keine neuen antimikrobiellen Wirkstoffe zur Behandlung gram-negativer Infektionen auf den Markt gekommen sind und in absehbarer Zeit auch nicht damit gerechnet werden kann.
Kontakt:
Dr. rer. nat. Jan Weile und
Prof. Dr. Cornelius Knabbe
Robert Bosch Krankenhaus
Labormedizin
D-Stuttgart
Tel.: 0711/8101-3501
Fax: 0711/8101-3618
cornelius.knabbe@rbk.de
jan.weile@ikp-stuttgart.de
www.rbk.de